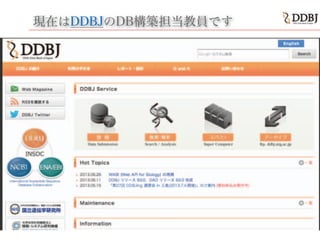
[DDBJing30] DDBJ と NIG SuperComputer の紹介、大量配列情報
- 1. DDBJ, NIG SuperComputer, 大量配列情報解析 DDBJセンター 中村保一 第30回 DDBJing 講習会 in 東京 (2014.12.18)
- 3. 植物関係のゲノム解析+DB屋でした ?著作 http://genome.microbedb.jp/cyanobase/! ! 光合成細菌のゲノム解析+データベース ?著作 The Arabidopsis Genome Initiative (2000)! Analysis of the genome sequence of the ?owering plant Arabidopsis thaliana.! Nature, 408, 796-815.! ! シロイヌナズナゲノムプロジェクトで! 全体の 1/4 (27 Mb, 6200 genes) の領域解析
- 6. 国際塩基配列データベースの一員 International Nucleotide Sequence Databank CollaborationDatabank Collaboration
- 7. DDBJ (from Release note 92) Jun Mashima, Hideo Aono, Yuji Ashizawa, Yukino Dobashi, Mayumi Ejima, Masahiro Fujimoto, Asami Fukuda, Tomohiro Hirai, Fumie Hirata, Naofumi Ishikawa, Toshikazu Katsumata, Chiharu Kawagoe, Shingo Kawahara, Yuichi Kodama, Junko Kohira, Takehide Kosuge, Kyungbum Lee, Mika Maki, Kimiko Mimura, Takeshi Moriyama, Yoshihisa Munakata, Naoko Murakata, Keiichi Nagai, Toshihisa Okido, Yoshihiro Okuda, Katsunaga Sakai, Makoto Sato, Yoshihiro Serizawa, Aimi Shiida, Yukie Shinyama, Rie Sugita, Kimiko Suzuki, Daisuke Takagi, Daisuke Takai, Haru Tsutsui, Koji Watanabe, Tomohiko Yasuda, Shigeru Yatsuzuka, Emi Yokoyama, Eli Kaminuma, Osamu Ogasawara, Kosaku Okubo, Toshihisa Takagi, and Yasukazu Nakamura ! ENA (from Release note 115) Blaise Alako, Clara Amid, Lawrence Bower, Ana Cerdeno-Taraga, Iain Cleland, Richard Gibson, Neil Goodgame, Petra ten Hoopen, Mikyung Jang, Simon Kay, Rasko Leinonen, Xin Liu, Arnaud Oisel, Rodrigo Lopez, Hamish McWilliam, Nima Pakseresht, Sheila Plaister, Rajesh Radhakrishnan, Kethy Reddy, Stephane Riviere, Marc Rossello, Nicole Silvester, Dmitriy Smirnov, Ana Luisa Toribio, Daniel Vaughan, Vadim Zalunin and Guy Cochrane ! GenBank (from Release note 195) Mark Cavanaugh, Ilene Mizrachi, Yiming Bao, Michael Baxter, Lori Black, Larissa Brown, Vincent Calhoun, Larry Chlumsky, Karen Clark, Jianli Dai, Michel Eschenbrenner, Irene Fang, Michael Fetchko, Linda Frisse, Andrea Gocke, Anjanette Johnston, Mark Landree, Jason Lowry, Suzanne Mate, Richard McVeigh, DeAnne Olsen Cravaritis, Leigh Riley, Susan Schafer, Beverly Underwood, Melissa Wright, Linda Yankie, Serge Bazhin, Evgueni Belyi, Colleen Bollin, Mark Cavanaugh, Yoon Choi, Ilya Dondoshansky, J. Bradley Holmes, WonHee Jang, Jonathan Kans, Leonid Khotomliansky, Michael Kimelman, Michael Kornbluh, Jim Ostell, Denis Sinyakov, Karl Sirotkin, Vladimir Soussov, Elena Starchenko, Hanzhen Sun, Tatiana Tatusova, Lukas Wagner, Eugene Yaschenko, Sergey Zhdanov, Slava Khotomliansky, Igor Lozitskiy, Craig Oakley, Eugene Semenov, Ben Slade, Constantin Vasilyev, Peter Cooper, Hanguan Liu, Wayne Matten, Scott McGinnis, Rana Morris, Steve Pechous, Monica Romiti, Eric Sayers, Tao Tao, Majda Valjavec-Gratian and David Lipman
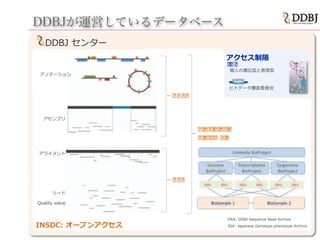
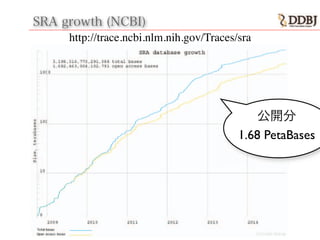
- 8. DDBJDDBJが運営しているデータベース DRA: DDBJ Sequence Read Archive JGA: Japanese Genotype-phenotype Archive
- 9. DDBJ登録ファイルの例 LOCUS AB091058 2109 bp DNA linear BCT 02-SEP-2003! DEFINITION Gluconacetobacter xylinus cmcase, ccp genes for! endo-beta-1,4-glucanase, cellulose complementing protein, complete! cds.! ACCESSION AB091058! VERSION AB091058.1! KEYWORDS .! SOURCE Gluconacetobacter xylinus! ORGANISM Gluconacetobacter xylinus! Bacteria; Proteobacteria; Alphaproteobacteria; Rhodospirillales;! Acetobacteraceae; Gluconacetobacter.! REFERENCE 1 (bases 1 to 2109)! AUTHORS Kawano,S., Tajima,K., Uemori,Y., Yamashita,H., Erata,T.,! Munekata,M. and Takai,M.! TITLE Direct Submission! JOURNAL Submitted (28-AUG-2002) to the DDBJ/EMBL/GenBank databases.! Contact:Kenji Tajima! Hokkaido University, Graduate School of Engineering; N13W8,! Kita-ku, Sapporo, Hokkaido 060-8628, Japan! REFERENCE 2 ! AUTHORS Kawano,S., Tajima,K., Uemori,Y., Yamashita,H., Erata,T.,! Munekata,M. and Takai,M.! TITLE Cloning of Cellulose Synthesis Related Genes from Acetobacter! xylinum ATCC23769 and ATCC53582: Comparison of Cellulose Synthetic! Ability Between ATCC23769 and ATCC53582! JOURNAL Unpublished (2002)! COMMENT ! FEATURES Location/Qualifiers! source 1..2109! /db_xref="taxon:28448"! /mol_type="genomic DNA"! /note="synonym:Acetobacter xylinum"! /organism="Gluconacetobacter xylinus"! /strain="ATCC 53582"! CDS 10..1038! /codon_start=1! /gene="cmcase"! /product="endo-beta-1,4-glucanase"! /protein_id="BAC82540.1"! /transl_table=11! /translation="MSVMAAMGGAQVLSSTGAFADTAPDAVAQQWAIFRAKYLRPSGR! VVDTGNGGESHSEGQGYGMLFAASAGDLASFQSMWMWARTNLQHTNDKLFSWRFLKGH! QPPVPDKNNATDGDLLIALALGRAGKRFQRPDYIQDAMAIYGDVLNLMTMKAGPYVVL! MPGAVGFTKKDSVILNLSYYVMPSLLQAFDLTADPRWRQVMEDGIRLVSAGRFGQWRL! PPDWLAVNRATGALSIASGWPPRFSYDAIRVPLYFYWAHMLAPNVLADFTRFWNNFGA! NALPGWVDLTTGARSPYNAPPGYLAVAECTGLDSAGELPTLDHAPDYYSAALTLLVYI! ARAEETIK" CDS 1035..2096! /codon_start=1! /gene="ccp"! /product="cellulose complementing protein"! /protein_id="BAC82541.1"! /transl_table=11! /translation="MSASGSDEVAGGGQAGSPQDFQRVLRSFGVEGGQYSYRPFVDRS! FDVTGVPEAVERHFDQAEHDTAVEEQVTPAPQIAVAPPPPPVVPDPPAIVTETAPPPP! VVVSAPVTYEPPAAAVPAEPPVQEAPVQAAPVPPAPVPPIAEQAPPAAPDPASVPYAN! VAAAPVPPDPAPVTPAPQARVTGPNTRMVEPFSRPQVRTVQEGATPSRVPSRSMNAFP! RTSASSISERPVDRGVADEWSPVPKARLSPRERPRPGDLSFFFQGMRDTRDEKKFFPV! ASTRSVRSNVSRMTSMTKTDTNSSQASRPGSPVASPDGSPTMAEVFMTLGGRATELLS! PRPSLREALLRRRENEEES"! BASE COUNT 343 a 661 c 661 g 444 t! ORIGIN ! 1 cgttccttta tgtcggtcat ggcggcgatg ggaggggcgc aggtgctttc atccaccggt! 61 gcgttcgcag acaccgcccc cgatgcggtc gcgcagcaat gggccatctt ccgcgccaag! 121 tatcttcgtc ccagcggacg tgtcgtggat acgggcaatg gtggcgaatc ccatagtgag! 181 gggcagggct atggcatgct ctttgccgcg tcggcggggg accttgcgtc gttccagtcg! 241 atgtggatgt gggcgcgcac caacctgcag cataccaatg acaagctgtt ttcctggcgg! 301 ttcctcaagg ggcatcagcc cccggtgccc gacaagaaca atgccacaga tggcgacctg! 361 ctgatcgcgc ttgcgcttgg tcgtgcgggc aagcgtttcc agcgccccga ttacattcag! 421 gacgccatgg ccatttatgg cgatgtgctg aacctgatga cgatgaaggc gggaccgtat! 481 gtcgtcctca tgcccggtgc tgtcggcttt accaagaagg acagcgtgat cctcaacctg! 541 tcctattacg tcatgccctc gctgctgcag gcgttcgacc ttacggccga cccgcgctgg! 601 cgtcaggtga tggaagacgg gattcgcctt gtttccgccg gccgtttcgg gcagtggcgc! 661 ctgccccccg actggctggc ggtgaatcgc gccaccggtg cgctgtcgat cgcatcggga! 721 tggccgccgc gcttttccta tgatgcgatt cgggtgccgc tttattttta ttgggcgcat! 781 atgctggcgc cgaacgtgtt ggctgatttc acccgattct ggaataattt cggggctaat! 841 gccctgccag gatgggttga tctgacaaca ggggcgcgtt cgccgtacaa cgccccgcct! 901 ggatatcttg ctgttgccga atgcacgggg cttgattctg ccggggaact cccgacactg! 961 gatcatgcgc ccgattatta ttccgcagcg ttgacgctgc tcgtttacat cgcgcgggcg! 1021 gaggagacta taaagtgagt gcttcagggt ctgatgaggt ggctggggga gggcaggctg! 1081 gaagtccgca ggattttcag cgggtcctgc gttcttttgg tgtcgaaggt gggcagtatt! 1141 cctaccggcc gtttgttgac cgttcctttg atgtgacagg cgtgcccgag gctgttgaaa! 1201 ggcacttcga tcaggcggag catgacacgg cggttgagga gcaggtcact cccgcgccac! 1261 aaatcgcggt cgcaccgcca ccgccgccag tcgttcctga cccgcccgcc atcgtgacgg! 1321 aaaccgcgcc cccgccgcct gtcgtggtca gcgctccggt cacgtatgaa cccccggctg! 1381 ccgccgtgcc ggcagagcct cccgttcagg aagcccccgt gcaggcggcg ccggttcccc! 1441 ccgcgcctgt gcccccgatt gcggagcagg ctcctcccgc ggcgccggac ccggcatccg! 1501 tgccgtatgc gaacgtcgcg gcagcacccg ttccacctga tcccgcaccg gttacgcctg! 1561 cgccgcaggc gcgcgtgacg gggccgaaca cccgtatggt ggagcccttt tcccgcccgc! 1621 aggtccgcac ggtgcaggag ggggcaaccc cgtcacgtgt accttcgcgt tcaatgaacg! 1681 ctttcccccg cacatcagca tcgtccataa gtgagcgtcc ggtggacagg ggtgttgccg! 1741 atgaatggag tcctgttccg aaggcacgcc tcagcccgcg ggagcgtccg cgtcccggcg! 1801 atctgagctt tttctttcag gggatgcgcg acacccgtga tgaaaagaag ttctttcccg! 1861 tggcgtccac gcgatcagtt cgttctaatg tttccaggat gaccagcatg accaagacag! 1921 acacgaattc ctctcaggct tctcgtcccg gcagccccgt cgcctcgcct gatgggtcgc! 1981 ccacaatggc cgaagtgttc atgacgctgg gtggtcgtgc gacggaactc ctcagccccc! 2041 gtccttcgct gcgggaggcg ctgttgcgtc gtcgtgaaaa cgaagaagaa tcctaaggcc! 2101 ctatattca! //! ! !
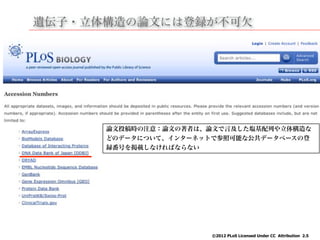
- 10. 遺伝子?立体構造の論文には登録が不可欠 ?2012 PLoS Licensed Under CC Attribution 2.5 論文投稿時の注意:論文の著者は、論文で言及した塩基配列や立体構造な どのデータについて、インターネットで参照可能な公共データベースの登 録番号を掲載しなければならない
- 11. INSDCに多くの配列が登録された生物種 DDBJに登録されている生物種 Top 100の ワードクラウド(数が多いほど大きい字で 表示) Images created by the Wordle.net web application are licensed under a Creative Commons Attribution 3.0 United States License. ヒト トウモロコシ マウス ラット ブタ ウシ
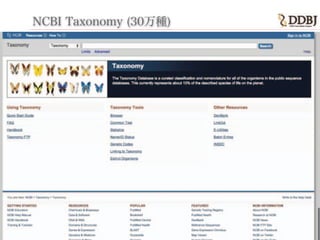
- 12. NCBI Taxonomy (30万種NCBI Taxonomy (30万種)
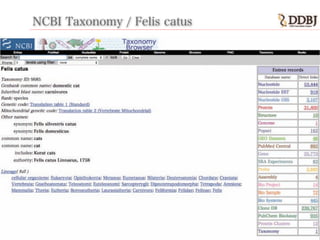
- 13. NCBI Taxonomy / Felis catus
- 14. 生物学の! 情報爆発
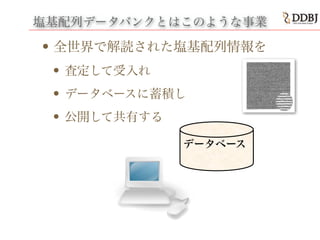
- 17. 「環境」シーケンス ?特定の環境からサンプリングした生物相のDNA を、培養することなく全解析することができる ? MetaGenomics Metagenomics is the study of metagenomes, genetic material recovered directly from environmental samples. The broad ?eld may also be referred to as environmental genomics, ecogenomics or community genomics. (by Wikipedia)
- 20. 代表的 NGS 機材 (左)Roche (454): GS FLX+ System (中)illumina: Genome Analyzer IIx System (右)Life Technologies: 5500 xl SOLiD System
- 22. NGSの例: illumina: GA の原理 フラットな固層上に適当な間隔でDNAを1分子ずつ 固定、基盤上で「ブリッジPCR」を行い、スポット としてDNAを増幅 相補鎖合成を行いながら化学発光をとらえる 4つの塩基に別々の蛍光標識をつけておいて、結合 した塩基の場所をスポットの光として特定し、塩 基配列を解読していく 元データは時系列の高密度な画像データ http://www.youtube.com/watch?v=77r5p8IBwJk
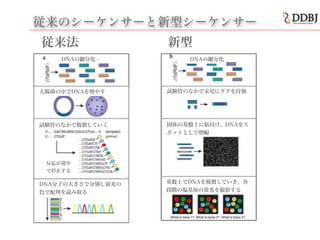
- 23. 新型シーケンサはなぜ高速?→「集積度」 ? 従来法は溶液やゲル中での反応と分離 ? 固体担体を用いて超高密度化を可能にした マイクロプレート 24 16 = 384穴 イルミナ社 GA フローセル 数千万スポット
- 24. さらに「ポータブル」シーケンサ
- 25. DRA!! DDBJ Sequence Read Archive
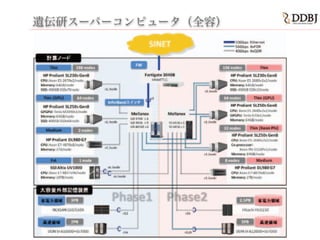
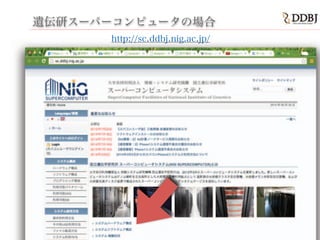
- 27. 遺伝研スーパー コンピュータ
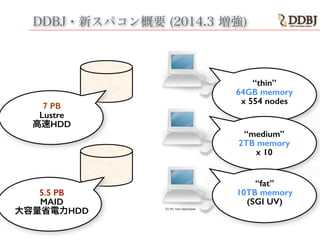
- 31. DDBJ?新スパコン概要 (2014.3 増強) 5.5 PB MAID 大容量省電力HDD 7 PB Lustre 高速HDD “medium” 2TB memory x 10 “thin” 64GB memory x 554 nodes “fat” 10TB memory (SGI UV) CC-PD from OpenClipart
- 32. 電子式計算機 ? 1946年 ENIAC ? Electronic Numerical Integrator and Calculator ? 「世界最初のコンピュータ」と目される ? 約 18,000 本の真空管からなる ? 10進法を採用 歯車式計算機の電子版 ? プログラム内蔵式 ではない http://commons.wikimedia.org/wiki/File:Eniac.jpg
- 33. プログラム内蔵電子式計算機:ノイマン型 ? 1949年 EDSAC(世界初の実用型) ? Electronic Delay Storage??????? Automatic Calculator ? 1951年 EDVAC (ENIAC後継) ? Electronic Discrete Variable??????? Automatic Computer ? 二進数を使用 ? メモリは 遅延記憶装置 を使用 EDSAC EDVAC
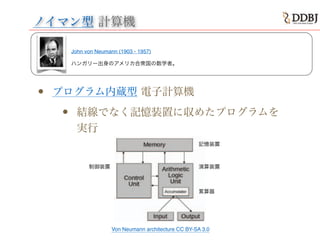
- 34. ノイマン型 計算機 ? プログラム内蔵型 電子計算機 ? 結線でなく記憶装置に収めたプログラムを 実行 John von Neumann (1903 - 1957)! ! ハンガリー出身のアメリカ合衆国の数学者。 Von Neumann architecture CC BY-SA 3.0 制御装置 演算装置 記憶装置 累算器
- 35. ノイマン型 計算機 ? プログラム内蔵型 電子計算機 ? 計算機のプログラムをコードで表し、数値 データと同じ様に記憶装置に記憶させる ? 一方、電気機械式計算機ではプログラムは 記憶されず、一連の命令として紙テープに穿 孔されていて、制御装置は順次テープから プログラムを読みだして実行する。 ? 命令が記憶装置に入っているので、演算を 行って計算している途中で命令を変更する ことができる。
- 36. UNIX Operating System のはじまり Bell 研の Ken Thompson, Dennis Ritchieらが Space Travel で遊ぶために(の目的だ けってわけでもないけど)高価で 買ってもらえなかった DEC-10 の かわりに部屋のスミに転がってい た借り物の PDP-7 で「えいやっ」 と作ってみた「小さくて」「軽い」 オペレーティングシステム (1968) Life with UNIX (アスキー出版局; 1990)より引用/改変
- 37. ところがどっこい iOS の基礎部分は Darwin で あり、NeXTSTEP を先祖に 持つ由緒正しき Berkeley Software Distribution (BSD) UNIX の系譜に連なります。 要するに組み込み系の UNIX (POSIX 準拠) で動作。 MacOS X も勿論 UNIX。 http://commons.wikimedia.org/wiki/File:IPhone_5.png
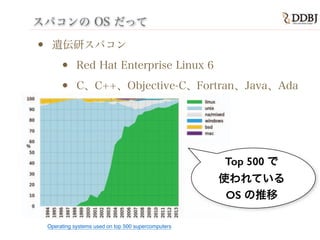
- 38. スパコンの OS だって ? 遺伝研スパコン ? Red Hat Enterprise Linux 6 ? C、C++、Objective-C、Fortran、Java、Ada Operating systems used on top 500 supercomputers Top 500 で 使われている OS の推移
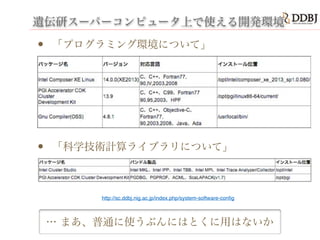
- 40. 遺伝研スーパーコンピュータ上で使える開発環境 ? 「プログラミング環境について」 ! ! ! ! ? 「科学技術計算ライブラリについて」 … まあ、普通に使うぶんにはとくに用はないか http://sc.ddbj.nig.ac.jp/index.php/system-software-con?g
- 41. シェル ? 「シェル環境について」 ? システムへのログインシェルは、bash, tcsh, zsh が利用可能です。 UNIX にコマンドを出すためのプログラムである 「シェル」は複数のなかから選べます。お好きな ものをどうぞ。 「シェルスクリプト」が書けるようになると、ぐっ と大量の解析作業が捗るようになりますよ。 http://sc.ddbj.nig.ac.jp/index.php/system-software-con?g
- 44. NGS s + SC s in Biology “medium” 2TB memory x 10 “fat” 10TB memory (SGI UV)
- 45. スパコン利用申請はこちら ?[ 遺伝研 スーパーコンピュータ ] で検索 無料です!
- 46. 遗伝子机能予测
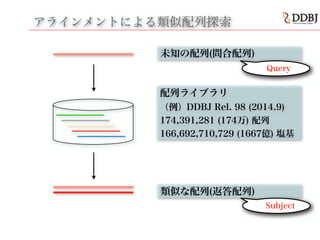
- 47. アラインメントによる類似配列探索 未知の配列(問合配列) 配列ライブラリ (例)DDBJ Rel. 98 (2014.9) 174,391,281 (174万) 配列 166,692,710,729 (1667億) 塩基 類似な配列(返答配列) 未知の配列(問合配列) Query 類似な配列(返答配列) Subject
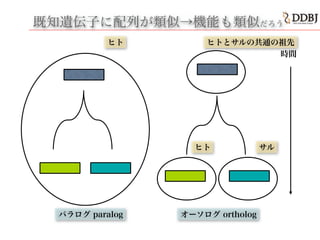
- 48. 既知遺伝子に配列が類似→機能も類似既知遺伝子に配列が類似→機能も類似だろう 時間 パラログ paralog オーソログ ortholog ヒト サル 時間 ヒトとサルの共通の祖先ヒト
- 49. 機能が実験的に予測されている遺伝子は酵母では 30% に達するが、シロイヌナズナでは 10% しか存在しな い。それ以外の遺伝子の機能注釈は、計算機の支援に よる「予測」にすぎない。 機能予測の根拠の割合 ※みなしご (Orphan) 遺伝子: 相同な配列の遺伝子が存在し ない遺伝子 「ゲノム2」MEDSi (2002) より
- 51. たとえばこんな配列があったとする “similar to Probable ubiquinone biosynthesis protein ubiB” >similar to Probable ubiquinone biosynthesis protein ubiB MSPAPMPVTTAEQDRDVVVIDAVVEEVRPPRLPKSHLEDLGPVSDMFPESWEYHPDLIME FYRKRPLQVLGRLINILFPLLRFILGIWWEKLRGKDPTVSRAKAIQLRELLTNLGPTYIK VGQALSTRPDLVPPVFLDELTTLQDQLPSFPNEVAYRFIEEELGAPAEEIYAELSPEPIA AASLGQVYKGKLKTGEAVAVKVQRPDLVRRITLDIYIMRSLSLWARRSVKRLRSDLVAIT DELASRVFEEMNYYQEAINGEKFAQLYGSLPEIYVPSIYWQYTGRRVLTMEWVEGIKLTN IKAIQAQGIDATHLVEVGVQCSLRQLLEHGFFHADPHPGNLLAMADGRLAYLDFGMMSTI QPYQRYGLIEAVVHLVNRDFDSLAKDYVKLDFLKPDTDLKPIIPALGQVFGNALGASVAE LNFKSITDQMSAMMYEFPFRVPAYYALIIRSMVTLEGIAIGIDPNFKVLSKAYPYIAKRL LTDQSEELRTSLKELLFKEGSFRWNRLENLLRNAKNSPGFDFDYVLNEATEFLLSDRGQF IRDRLVAELVNSIDQLGRNTWQQVSHNIQERISFLGDLGNGNGKAHQTKTIKVVPQPAIA QQEETWQHLQNLWQILKETPGFDPLKFVPVLSQIIVNPTSRRMGQQVAEGLLQKAIARVI RQWALALESQPNPAIKIRNAA !
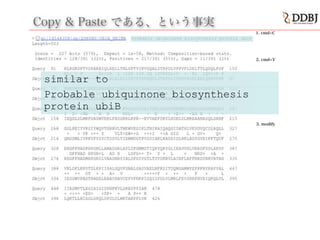
- 52. Copy & Paste である、という事実 1. cmd+C Probable ubiquinone biosynthesis protein ubiB 2. cmd+V 3. modify similar to
- 53. 機能 annotation 完成! >similar to Probable ubiquinone biosynthesis protein ubiB MSPAPMPVTTAEQDRDVVVIDAVVEEVRPPRLPKSHLEDLGPVSDMFPESWEYHPDLIME FYRKRPLQVLGRLINILFPLLRFILGIWWEKLRGKDPTVSRAKAIQLRELLTNLGPTYIK VGQALSTRPDLVPPVFLDELTTLQDQLPSFPNEVAYRFIEEELGAPAEEIYAELSPEPIA AASLGQVYKGKLKTGEAVAVKVQRPDLVRRITLDIYIMRSLSLWARRSVKRLRSDLVAIT DELASRVFEEMNYYQEAINGEKFAQLYGSLPEIYVPSIYWQYTGRRVLTMEWVEGIKLTN IKAIQAQGIDATHLVEVGVQCSLRQLLEHGFFHADPHPGNLLAMADGRLAYLDFGMMSTI QPYQRYGLIEAVVHLVNRDFDSLAKDYVKLDFLKPDTDLKPIIPALGQVFGNALGASVAE LNFKSITDQMSAMMYEFPFRVPAYYALIIRSMVTLEGIAIGIDPNFKVLSKAYPYIAKRL LTDQSEELRTSLKELLFKEGSFRWNRLENLLRNAKNSPGFDFDYVLNEATEFLLSDRGQF IRDRLVAELVNSIDQLGRNTWQQVSHNIQERISFLGDLGNGNGKAHQTKTIKVVPQPAIA QQEETWQHLQNLWQILKETPGFDPLKFVPVLSQIIVNPTSRRMGQQVAEGLLQKAIARVI RQWALALESQPNPAIKIRNAA ! !
- 54. Copy & Paste による automatic な! 継承で! ゴミが蓄積される
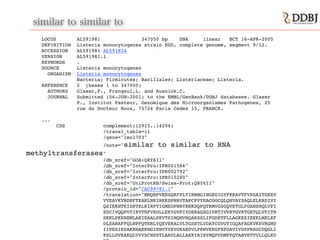
- 55. similar to similar to LOCUS AL591981 347050 bp DNA linear BCT 16-APR-2005! DEFINITION Listeria monocytogenes strain EGD, complete genome, segment 9/12.! ACCESSION AL591981 AL591824! VERSION AL591981.1! KEYWORDS .! SOURCE Listeria monocytogenes! ORGANISM Listeria monocytogenes! Bacteria; Firmicutes; Bacillales; Listeriaceae; Listeria.! REFERENCE 2 (bases 1 to 347050)! AUTHORS Glaser,P., Frangeul,L. and Rusniok,C.! JOURNAL Submitted (06-JUN-2001) to the EMBL/GenBank/DDBJ databases. Glaser ! P., Institut Pasteur, Genomique des Microorganismes Pathogenes, 25 ! rue du Docteur Roux, 75724 Paris Cedex 15, FRANCE. ! ! ...! CDS complement(12915..14294)! /transl_table=11! /gene="lmo1703"! /note="similar to similar to RNA methyltransferases"! /db_xref="GOA:Q8Y6I1"! /db_xref="InterPro:IPR001566"! /db_xref="InterPro:IPR002792"! /db_xref="InterPro:IPR010280"! /db_xref="UniProtKB/Swiss-Prot:Q8Y6I1"! /protein_id="CAC99781.1"! /translation="MNQNPVEEGQKFPLTIRRMGINGEGIGYFKKAVVFVPGAITGEEV! VVEAVKVRDRFTEAKLNKIRKKSPNRVTAPCPVYEACGGCQLQHVAYSAQLELKRDIVI! QSIEKHTKIDPTKLKIRPTIGMEDPWRYRNKSQFQTRMVGSGQVETGLFGANSHQLVPI! EDCIVQQPVTIKVTNFVRDLLEKYGVPIYDEKAGSGIVRTIVVRTGVKTGETQLVFITN! SKKLPKKREMLAEIEAALPEVTSIMQNVNQAKSSLIFGDETFLLAGKESIEEKLMELEF! DLSARAFFQLNPFQTERLYQEVEKALVLTGSETLVDAYCGVGTIGQAFAGKVKEVRGMD! IIPESIEDAKRNAEKNGIENVYYEVGKAEDVLPKWVKEGFRPDAVIVDPPRSGCDQGLI! KSLLDVEAKQLVYVSCNPSTLARDLALLAKKYRIRYMQPVDMFPQTAHVETVVLLQLKD!
- 56. Copy & paste error! >gi|91204169|emb|CAJ71822.1| strongly imilar to aspartate aminotransferase [Candidatus Kuenenia stuttgartiensis]! MIASRMSNIDSSGIRKVFDLAQKMKSPVNLSIGQPDFDVPGEIKEVAIKSINEGANKYTLTQGIPELRNV! ...! >gi|31541577|gb|AAP56877.1| predicted methyl transferas [Mycoplasma gallisepticum R]! MSALYLVGLPIGNLSEINHRALEILNQLEIIYCENTDNFKKLLNLLNINFRDKKLISYHKFNETNRFIMI! ... similar to! transferase similar to transferase
- 57. SEPT2 2-Sep case in Refseq LOCUS XM_392412 2125 bp mRNA linear INV 12-APR-2011! DEFINITION PREDICTED: Apis mellifera septin-2 (2-Sep), mRNA.! ACCESSION XM_392412! VERSION XM_392412.4 GI:328785636! KEYWORDS .! SOURCE Apis mellifera (honey bee)! ORGANISM Apis mellifera! Eukaryota; Metazoa; Arthropoda; Hexapoda; Insecta; Pterygota;! Neoptera; Endopterygota; Hymenoptera; Apocrita; Aculeata; Apoidea;! Apidae; Apis.! COMMENT MODEL REFSEQ: This record is predicted by automated computational! analysis. This record is derived from a genomic sequence! (NW_003378075) annotated using gene prediction method: GNOMON,! supported by EST evidence.! Also see:! Documentation of NCBI's Annotation Process! ! On Apr 12, 2011 this sequence version replaced gi:110757583.! FEATURES Location/Qualifiers! source 1..2125! /organism="Apis mellifera"! /mol_type="mRNA"! /strain="DH4"! /db_xref="taxon:7460"! /linkage_group="LG6"! gene 1..2125! /gene="2-Sep"! /note="Derived by automated computational analysis using! gene prediction method: GNOMON. Supporting evidence! includes similarity to: 436 ESTs, 11 Proteins"! /db_xref="BEEBASE:GB17411"! /db_xref="GeneID:408882"! misc_feature 164..166! /gene="2-Sep"! /note="upstream in-frame stop codon"! CDS 194..1444! /gene="2-Sep"! /codon_start=1! /product="septin-2"! /protein_id="XP_392412.2" http://www.biomedcentral.com/1471-2105/5/80 http://www.ncbi.nlm.nih.gov/nuccore/XM_392412 LOCUS XM_392412 2125 bp mRNA linear INV 12-APR-2011 「セプチン2」 遺伝子が 「9月2日」に
- 58. contains similarity ? automated annotation: contains similarity to DNA-binding protein DNA binding domain “similar” region DNA binding protein unknown protein ↑without the domain!
- 59. どうすれば良いのか? ? 配列類似検索の対象は、信頼で きるライブラリから順に使う ? 配列類似検索以外の機能予測方 法を用いる ? 注釈の「根拠 (evidence)」が明 示できる方法で注釈する
- 60. どうすれば良いのか? ? 配列類似検索の対象は、信頼で きるライブラリから順に使う ? 配列類似検索以外の機能予測方 法を用いる ? 注釈の「根拠 (evidence)」が明 示できる方法で注釈する
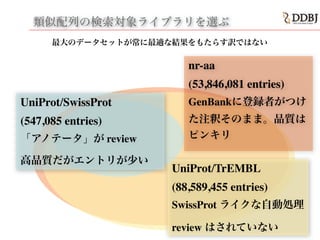
- 61. 類似配列の検索対象ライブラリを選ぶ 最大のデータセットが常に最適な結果をもたらす訳ではない UniProt/TrEMBL! (88,589,455 entries)! SwissProt ライクな自動処理! review はされていない UniProt/SwissProt! (547,085 entries)! 「アノテータ」が review 高品質だがエントリが少い nr-aa! (53,846,081 entries)! GenBankに登録者がつけ た注釈そのまま。品質は ピンキリ
- 62. どうすれば良いのか? ? 配列類似検索の対象は、信頼で きるライブラリから順に使う ? 配列類似検索以外の機能予測方 法を用いる ? 注釈の「根拠 (evidence)」が明 示できる方法で注釈する
- 66. InterPro でまとめがけ ?機能と構造単位の諸検索法の統合 ?モチーフ?ドメイン?プロファイル 等の名称で呼ばれる機能や構造と結 びついたアミノ酸配列の保存された 領域をまとめたデータベース ?Pfam, PRINTS, PROSITE... 等を総合 し、独自注釈も追加
- 68. どうすれば良いのか? ? 配列類似検索の対象は、信頼で きるライブラリから順に使う ? 配列類似検索以外の機能予測方 法を用いる ? 注釈の「根拠 (evidence)」が明 示できる方法で注釈する
- 69. IDA! Inferred from Direct Assay TAS! Traceable Author Statement ISS! Inferred from Sequence or Structural similarity Evidence codes in GO Annotation IEA! Inferred from Electronic Annotation (automated) IMP! Inferred from Mutant Phenotype
- 71. 正確な! 解析情報を! 付与して












![NGS!!
[次世代]Next-Generation Sequencer!
?!
[新型]New Generation Sequencer](https://image.slidesharecdn.com/30ddbjingddbj-141224194318-conversion-gate02/85/DDBJing30-DDBJ-NIG-SuperComputer-19-320.jpg)













![スパコン利用申請はこちら
?[ 遺伝研 スーパーコンピュータ ] で検索
無料です!](https://image.slidesharecdn.com/30ddbjingddbj-141224194318-conversion-gate02/85/DDBJing30-DDBJ-NIG-SuperComputer-45-320.jpg)






![Copy & paste error!
>gi|91204169|emb|CAJ71822.1| strongly imilar to aspartate
aminotransferase [Candidatus Kuenenia stuttgartiensis]!
MIASRMSNIDSSGIRKVFDLAQKMKSPVNLSIGQPDFDVPGEIKEVAIKSINEGANKYTLTQGIPELRNV!
...!
>gi|31541577|gb|AAP56877.1| predicted methyl transferas
[Mycoplasma gallisepticum R]!
MSALYLVGLPIGNLSEINHRALEILNQLEIIYCENTDNFKKLLNLLNINFRDKKLISYHKFNETNRFIMI!
...
similar to!
transferase
similar to
transferase](https://image.slidesharecdn.com/30ddbjingddbj-141224194318-conversion-gate02/85/DDBJing30-DDBJ-NIG-SuperComputer-56-320.jpg)