Modellistica molecolare e applicazioni alla sclerosi multipla
- 1. Enrico Pieroni ep@crs4.it 27 Maggio 2015 Modellistica molecolare e applicazioni alla sclerosi multipla
- 2. Outline ’ü¼ Cos'├© un modello? ’ü¼ Cos'├© una simulazione? ’ü¼ L'importanza delle scale temporali e spaziali ’ü¼ Il riconoscimento antigenico ’ü¼ La sclerosi multipla ’ü¼ Il mimetismo molecolare ’ü¼ Riconoscimento molecolare in sclerosi multipla ’ü¼ Risultati delle simulazioni sulle proteine collegate alla sclerosi multipla
- 3. Modello Situazione ŌĆ£realeŌĆØ. Modello: concettualizzazione, semplificazione della realt├Ā, spesso con formalismo matematico (grafo) ... che cattura aspetti essenziali (connettivit├Ā) e elimina quelli non rilevanti (distanze, geome_ tria, direzioni, ...) per gli scopi che ci si prefigge [sele_ zione caratteristiche, gradi di libert├Ā]. Idea: Markus J. Buehler (MIT)
- 4. Simulazione Cosa succederebbe se cavalcassi un raggio di luce? (A. Einstein, 1905)
- 5. Simulazione Soluzione (delle equazioni) del modello. Rappresenta una sorta di esperimento virtuale. Perch├© simulare? Gli esperimenti talvolta sono impossibili, costosi, pericolosi, richiedono tempi lunghi, alcune scale temporali e spaziali di interesse non sono accessibili, ... Inoltre, la simulazione consente di: interpretare i dati ŌĆ£sperimentaliŌĆØ, aiutare a capire i meccanismi sottostanti e predire nuove propriet├Ā del sistema reale, suggerire/selezionare esperimenti, aiutarne la progettazione, ... La simulazione offre un quadro dinamico, dove ├© possibile cambiare i parametri del sistema alla ricerca di propriet├Ā emergenti (sistemiche).
- 6. Simulazione al Calcolatore Oggi la simulazione avviene spesso tramite computer, ... e la velocit├Ā di simulazione scala con la potenza computazionale! Per cui oggi ├© possibile simulare sistemi complessi, quindi interessanti, quali i sistemi biologici. Blue Gene 2011, circa 700 TeraFLOPS: 700 000 000 000 000 op./s 1 giorno ENIAC 1946, 5 000 operazioni/s circa 2.5 milioni di secoli
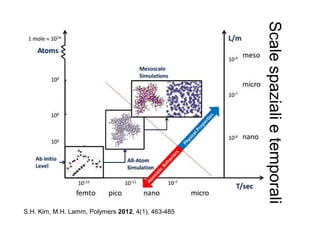
- 7. Sistemi a diverse scale L-T Elaborazione da idea di Aatto Laaksonen (Stokholm U.) ps ns ╬╝s fs m = 1 / 1 000 ╬╝ = 1 / 1 000 000 n = 1 / 1 000 000 000 p = 1 / 1 000 000 000 000 f = 1 / 1 000 000 000 000 000 psps 1 ├ģ Cristallografia a raggi X, spettroscopia NMR,
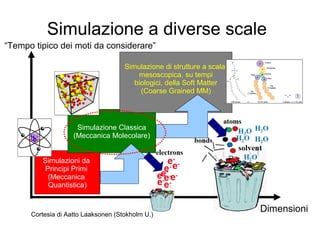
- 8. Simulazione a diverse scale Simulazioni da Principi Primi (Meccanica Quantistica) Simulazione Classica (Meccanica Molecolare) Simulazione di strutture a scala mesoscopica, su tempi biologici, della Soft Matter (Coarse Grained MM) Cortesia di Aatto Laaksonen (Stokholm U.) Dimensioni ŌĆ£Tempo tipico dei moti da considerareŌĆØ
- 9. ╬╝icro ŌĆō Macroscopico ’ü¼ Simulazione molecolare: genera tante configurazioni del sistema, misura le propriet├Ā statistiche e (tramite la fisica statistica) le collega a quelle macroscopiche ’ü¼ Alcuni parametri importanti: ’ü¼ Scala temporale dei moti inclusi nel modello (condiziona il passo di risoluzione delle equazioni) ’ü¼ Scala temporale degli effetti osservabili di interesse su scala macroscopica (condiziona il tempo totale di simulazione) ’ü¼ Dimensione del sistema (condiziona il tempo totale di simulazione) Comportamento ╬╝: moto atomico e molecolare, forze interatomiche ed intermolecolari Comportamenti M osservabili: propriet├Ā termodinamiche (T,P,V), di trasporto (velocit├Ā di diffusione), ottiche, ... Comportamento M bio: Legame, modifiche strutturali, attivazione recettore, diffusione farmaci attraverso membrana, ...
- 10. Applicazioni della Modellistica Molecolare Struttura delle proteine, per omologia con strutture cristallografiche. Interazione Proteina:Molecola, ad esempio Farmaco:Recettore. Interazione Proteina:Peptide, ad esempio in segnalazione e regolazione cellulare, riconoscimento degli antigeni (p:MHC). Proteina:Proteina ad es. pMHC:TCR, metabolismo, segnalazione cellulare, funzionamento muscolare. Proteina:Lipide, ad es. Recettore:Lipide, segnalazazione cellulare, sistema immunitario, sindrome metabolica. Proteina:Carboidrato, ad es. in infiammazioni, riconoscimento patogeni, adesione e segnalamento cellulare. Proteina:Polimero, ad esempio per il trasporto dei farmaci.
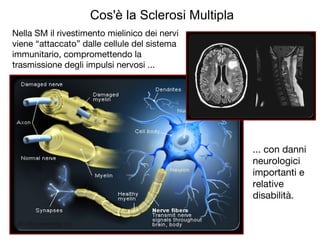
- 11. Nella SM il rivestimento mielinico dei nervi viene ŌĆ£attaccatoŌĆØ dalle cellule del sistema immunitario, compromettendo la trasmissione degli impulsi nervosi ... ... con danni neurologici importanti e relative disabilit├Ā. Cos'├© la Sclerosi Multipla
- 12. Fattori Genetici Evidenza: Gemelli monozigotici: 25-30% Gemelli dizigotici: 3-4% MHC (HLA): principale determinante genetica Quali sono le cause? Fattori Ambientali (sconosciuti!) -Esposizione solare (Vit. D), -Agenti patogeni, part. virus di Epstein-Barr (EBV), Micobatterio MAP -Fumo. Cocco, E., Sardu, C., Pieroni, E., et al. 2012 PLoS One International Multiple Sclerosis Genetics, C., Wellcome Trust Case Control, Sawcer, S.,et al. 2011 Nature Inaugurazione AA 2014-2015 Prolusione della Prof.ssa MG Marrosu, ŌĆ£Immunit├Ā in Sardegna: dono degli dei o mela avvelenata?ŌĆØ http://www.unica.it/UserFiles/File/Utenti/verdeoro/2014/12/prolusione2014_marrosu.pdf
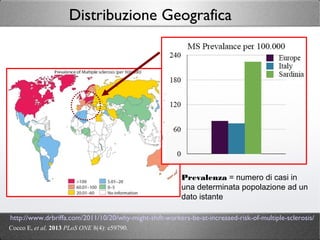
- 13. http://www.drbriffa.com/2011/10/20/why-might-shift-workers-be-at-increased-risk-of-multiple-sclerosis/ Cocco E, et al. 2013 PLoS ONE 8(4): e59790. Distribuzione Geografica Prevalenza = numero di casi in una determinata popolazione ad un dato istante
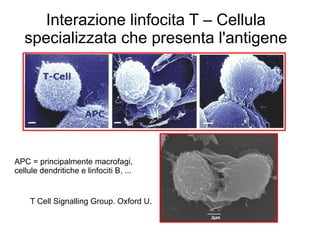
- 14. T Cell Signalling Group. Oxford U. Interazione linfocita T ŌĆō Cellula specializzata che presenta l'antigene APC = principalmente macrofagi, cellule dendritiche e linfociti B, ...
- 15. CMAJ July 18, 2006 vol. 175 no. 2 165-170 TCR : [peptide : MHC] Ecco qua il nostro attore principale Peptide o Antigene
- 16. PDB 3RDT Crystal Structure of 809.B5 TCR + MHC Class II I-Ab/3k peptide TCR : [peptide : MHC] Struttura Cristallografica T Cell peptide MHC Tasca di legame
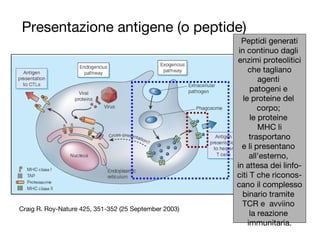
- 17. Presentazione antigene (o peptide) Craig R. Roy-Nature 425, 351-352 (25 September 2003) Peptidi generati in continuo dagli enzimi proteolitici che tagliano agenti patogeni e le proteine del corpo; le proteine MHC li trasportano e li presentano all'esterno, in attesa dei linfo- citi T che riconos- cano il complesso binario tramite TCR e avviino la reazione immunitaria.
- 18. La Dinamica Molecolare HLA-DRB1:1501 - EBV La DM ├©, in pratica, un filmino, con tante informazioni visive sulle configurazioni del complesso, che vanno poi quantificate attraverso l'analisi statistica di tutti i fotogrammi.
- 19. Investigare la plasticit├Ā del sistema peptide- MHC, confrontare i risultati per peptidi del proprio corpo e provenienti da agenti patogeni, correlare le caratteristiche strutturali e dinamiche del complesso peptide-MHC alla suscettibilit├Ā verso la malattia, utilizzando tecniche di Dinamica Molecolare. Il nostro Obiettivo
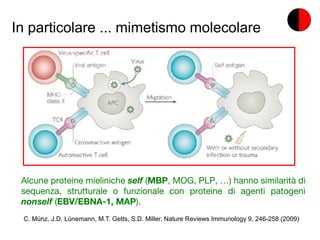
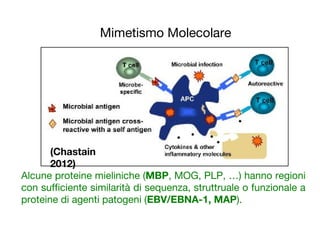
- 20. In particolare ... mimetismo molecolare C. M├╝nz, J.D. L├╝nemann, M.T. Getts, S.D. Miller, Nature Reviews Immunology 9, 246-258 (2009) Alcune proteine mieliniche self (MBP, MOG, PLP, ŌĆ”) hanno similarit├Ā di sequenza, strutturale o funzionale con proteine di agenti patogeni nonself (EBV/EBNA-1, MAP).
- 21. 2012 Si parte dalla Genomica
- 22. Le prime simulazioni molecolari
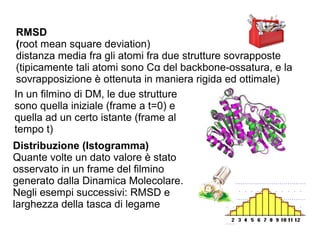
- 23. RMSD (root mean square deviation) distanza media fra gli atomi fra due strutture sovrapposte (tipicamente tali atomi sono C╬▒ del backbone-ossatura, e la sovrapposizione ├© ottenuta in maniera rigida ed ottimale) In un filmino di DM, le due strutture sono quella iniziale (frame a t=0) e quella ad un certo istante (frame al tempo t) Distribuzione (Istogramma) Quante volte un dato valore ├© stato osservato in un frame del filmino generato dalla Dinamica Molecolare. Negli esempi successivi: RMSD e larghezza della tasca di legame
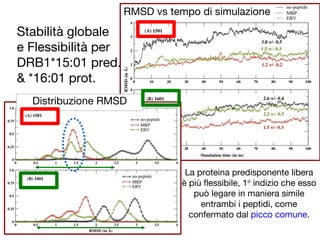
- 24. Stabilit├Ā globale e Flessibilit├Ā per DRB1*15:01 pred. & *16:01 prot. La proteina predisponente libera ├© pi├╣ flessibile, 1o indizio che esso pu├▓ legare in maniera simile entrambi i peptidi, come confermato dal picco comune. RMSD vs tempo di simulazione Distribuzione RMSD
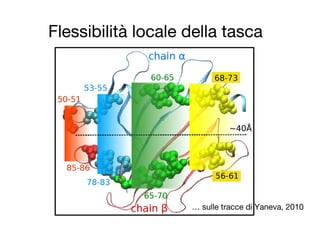
- 25. Flessibilit├Ā locale della tasca ŌĆ” sulle tracce di Yaneva, 2010
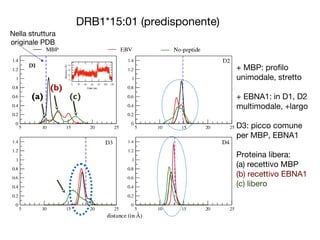
- 26. DRB1*15:01 (predisponente) (a) (b) (c) Nella struttura originale PDB + MBP: profilo unimodale, stretto + EBNA1: in D1, D2 multimodale, +largo D3: picco comune per MBP, EBNA1 Proteina libera: (a) recettivo MBP (b) recettivo EBNA1 (c) libero
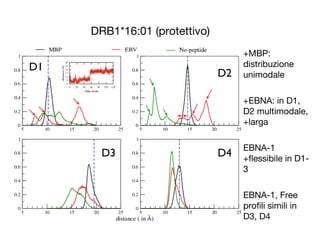
- 27. DRB1*16:01 (protettivo) +MBP: distribuzione unimodale +EBNA: in D1, D2 multimodale, +larga EBNA-1 +flessibile in D1- 3 EBNA-1, Free profili simili in D3, D4 D1 D2 D4D3
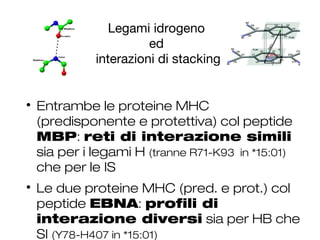
- 28. Legami idrogeno ed interazioni di stacking ’ü¼ Entrambe le proteine MHC (predisponente e protettiva) col peptide MBP: reti di interazione simili sia per i legami H (tranne R71-K93 in *15:01) che per le IS ’ü¼ Le due proteine MHC (pred. e prot.) col peptide EBNA: profili di interazione diversi sia per HB che SI (Y78-H407 in *15:01)
- 29. Energie di legame ... confermano che i) la proteina predisponente lega il peptide self ed il nonself in maniera simile, e che ii) il contrario vale per la proteina protettiva.
- 30. Primo ŌĆ£take home messageŌĆØ ’ü¼ La proteina MHC DR2 predisponente lega in maniera simile sia il peptide derivato da MBP che quello derivato da EBV, ... ’ü¼ ... mentre la proteina protettiva si lega pi├╣ debolmente al peptide virale e mostra configurazioni di legame nettamente distinte coi due peptidi. ’ü¼ Questo ├© in accordo con l'ipotesi di mimetismo molecolare: il peptide self (derivato dalla mielina) e quello nonself (virale) hanno somiglianza strutturale e funzionale nel legarsi alla
- 31. Supporto Sperimentale I pazienti EBV+ hanno risposta immunitaria anticorpale (mediata dai linfociti B) simile nei confronti dei peptidi EBNA1 ed MBP. Anticorpi reattivi contro il peptide MBP sono cross-reattivi contro il peptide EBNA1. EBNA1 ed MBP non hanno omologia di sequenza, quindi devono condividere omologia conformazionale quando sono presentati dalla proteina MHC.
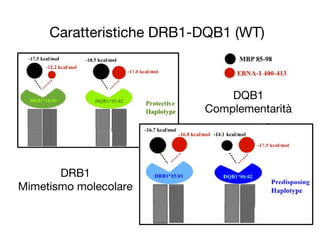
- 34. Caratteristiche DRB1-DQB1 (WT) DRB1 Mimetismo molecolare DQB1 Complementarit├Ā
- 35. Altre analisi ... Un breve accenno: l'analisi delle reti di interazione idrogeno e di stacking, le distribuzioni di larghezza locale della tasca di legame, sia sulle proteine WT che su quelle mutanti, mostrano i dettagli del processo molecolare di riconoscimento da parte delle proteine DRB1 e DQB1, in particolare della specificit├Ā relativamente al tipo di antigene (self/nonself) e di tessuto, e nel loro complesso confermando l'ipotesi di
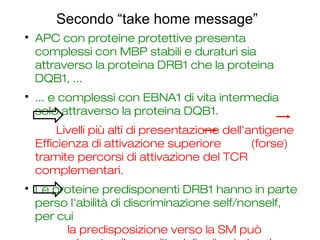
- 36. Secondo ŌĆ£take home messageŌĆØ ’ü¼ APC con proteine protettive presenta complessi con MBP stabili e duraturi sia attraverso la proteina DRB1 che la proteina DQB1, ... ’ü¼ ... e complessi con EBNA1 di vita intermedia solo attraverso la proteina DQB1. Livelli pi├╣ alti di presentazione dell'antigene Efficienza di attivazione superiore (forse) tramite percorsi di attivazione del TCR complementari. ’ü¼ Le proteine predisponenti DRB1 hanno in parte perso l'abilit├Ā di discriminazione self/nonself, per cui la predisposizione verso la SM pu├▓
- 37. Terzo ŌĆ£take home messageŌĆØ Le proteine DRB e DQB sembrano avere un sistema di riconoscimento complementare e specifico a seconda del tessuto. Dato che: i) l'espressione delle proteine DRB1 e DQB1 ├© dipendente dal tessuto e ii) il mimetismo molecolare richiede una attivazione periferica dei linfociti T prima della riattivazione dei linfociti autoreattivi a livello del sistema nervoso centrale .... ’ü¼ Il ruolo della proteina DRB1 potrebbe essere maggiormente rilevante nella fase di attivazione periferica, ossia di ŌĆ£incontroŌĆØ iniziale con il fattore scatenante ambientale, mentre ... ’ü¼ ... il ruolo della proteina DQB1 potrebbe essere fondamentale nella riattivazione periferica del repertorio di linfociti T clonalmente espansi a partire dal primo incontro, che andrebbero poi ŌĆ£all'attaccoŌĆØ della mielina.
- 38. Supporto sperimentale del ruolo DRB/DQB
- 39. Supporto sperimentale del ruolo DRB/DQB
- 40. Progetto di piccoli peptidi ... che modulino l'affinit├Ā verso peptidi self/nonself, tramite l'aumento o la soppressione del carico del peptide nella tasca MHC, ... che agiscano selettivamente rispetto ai tessuti (e quindi alle fasi di arruolamento erroneo dei linfociti T). Integrazione di dati fenotipici, clinici, medici, genetici, metabolici e di modelistica, al fine di realizzare un modello individuale di rischio nei confronti della malattia e personalizzare il trattamento terapeutico. Potenzialit├Ā in Terapia
- 41. Conclusioni La modellistica computazionale a scala molecolare consente di: ’ü¼ Integrare le informazioni genetiche e ŌĆ£sperimentaliŌĆØ; ’ü¼ Esplorare potenziali meccanismi funzionali nella SM;< In prospettiva: ’ü¼ Aiutare il progetto di molecole e peptidi terapeutici; ’ü¼ Suggerire nuovi ŌĆ£esperimentiŌĆØ. Come ├© tipico per i sistemi biologici, non abbiamo una teoria generale, quanto visto si applica a determinate proteine (pur collegate alla malattia) e determinati peptidi, occorreranno altri esperimenti,
- 43. (Modeling) Limits/Perspectives ’ü¼ MS: + alleles, + peptides, TCR; ’ü¼ FMS: less relevant lysoPCs, in general interaction of molecules diet-derived with biological processes; ’ü¼ CASQ2: cardiac isoform; ’ü¼ Therapeutical peptides, tissue- and antigen-selective, modulating autoimmune response; ’ü¼ Molecules interfering with PAFR & modulating pain response; ’ü¼ FMS bio-markers from metabolic profiles: ’ü¼ Clinical and phenotypical data integration; ’ü¼ Individual Risk model, Prognosis and Drug response in MS;
- 44. Un nuovo potenziale antigene: il MAP
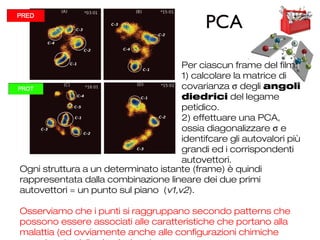
- 45. PCA Ogni struttura a un determinato istante (frame) ├© quindi rappresentata dalla combinazione lineare dei due primi autovettori = un punto sul piano (v1,v2). Osserviamo che i punti si raggruppano secondo patterns che possono essere associati alle caratteristiche che portano alla malattia (ed ovviamente anche alle configurazioni chimiche Per ciascun frame del film: 1) calcolare la matrice di covarianza Žā degli angoli diedrici del legame petidico. 2) effettuare una PCA, ossia diagonalizzare Žā e identifcare gli autovalori pi├╣ grandi ed i corrispondenti autovettori.
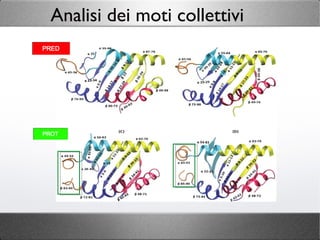
- 46. Analisi dei moti collettivi
- 47. Dinamica dell'acqua ed Energia di legame Ci sono pi├╣ molecole d'acqua attorno alle proteine protettive Valori di entropia maggiori per MAP che lega le proteine predisponenti
- 48. S.H. Kim, M.H. Lamm, Polymers 2012, 4(1), 463-485 Scalespazialietemporali
- 49. 4
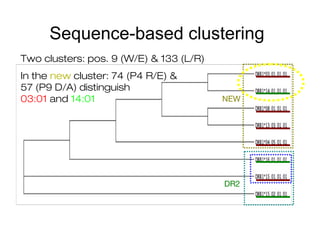
- 50. DR2 Sequence-based clustering Two clusters: pos. 9 (W/E) & 133 (L/R) In the new cluster: 74 (P4 R/E) & 57 (P9 D/A) distinguish 03:01 and 14:01 NEW
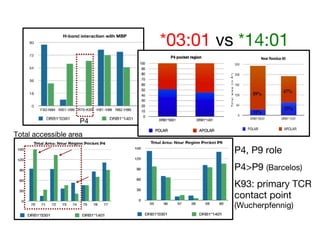
- 51. *03:01 vs *14:01 P4 P4, P9 role P4>P9 (Barcelos) K93: primary TCR contact point (Wucherpfennig) Total accessible area
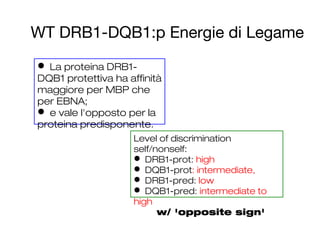
- 52. WT DRB1-DQB1:p Energie di Legame Level of discrimination self/nonself: ’ü¼ DRB1-prot: high ’ü¼ DQB1-prot: intermediate, ’ü¼ DRB1-pred: low ’ü¼ DQB1-pred: intermediate to high w/ 'opposite sign' ’ü¼ La proteina DRB1- DQB1 protettiva ha affinit├Ā maggiore per MBP che per EBNA; ’ü¼ e vale l'opposto per la proteina predisponente.
- 53. Discussion *03:01, *04:01: very similar by sequence but striking functional differences, particularly binding MBP. Synergistic interactive effect: resistance allele *14:01 binds MBP weaker at P9 and stronger at P4 pos. 70 than susceptibility allele *03:01. *03:01 shows a quite distinct P4 polar environment, respect to *14:01.
- 54. Mimetismo Molecolare Alcune proteine mieliniche (MBP, MOG, PLP, ŌĆ”) hanno regioni con sufficiente similarit├Ā di sequenza, struttruale o funzionale a proteine di agenti patogeni (EBV/EBNA-1, MAP). (Chastain 2012)
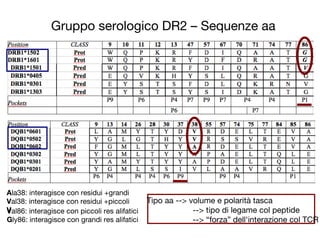
- 55. Gruppo serologico DR2 ŌĆō Sequenze aa Val86: interagisce con piccoli res alifatici Gly86: interagisce con grandi res alifatici Ala38: interagisce con residui +grandi Val38: interagisce con residui +piccoli V Tipo aa --> volume e polarit├Ā tasca --> tipo di legame col peptide --> ŌĆ£forzaŌĆØ dell'interazione col TCR
- 56. Gli attori/ici principali del ŌĆ£filmŌĆØ MBP(85-98) EBNA-1(400-413) Dimero con 2 catene A, B, ciascuna con 2 domini ╬▒, ╬▓ Residui specifici per ancoraggio e riconoscimento profili di legame del peptide



![Modello
Situazione ŌĆ£realeŌĆØ.
Modello:
concettualizzazione,
semplificazione della
realt├Ā, spesso con
formalismo matematico
(grafo) ... che cattura aspetti essenziali
(connettivit├Ā) e
elimina quelli non rilevanti
(distanze, geome_
tria, direzioni, ...)
per gli scopi che ci
si prefigge [sele_
zione caratteristiche, gradi di libert├Ā]. Idea: Markus J. Buehler (MIT)](https://image.slidesharecdn.com/pieroni-150724112126-lva1-app6892/85/Modellistica-molecolare-e-applicazioni-alla-sclerosi-multipla-3-320.jpg)






![CMAJ July 18, 2006 vol. 175 no. 2 165-170
TCR : [peptide : MHC]
Ecco qua il nostro
attore principale
Peptide o
Antigene](https://image.slidesharecdn.com/pieroni-150724112126-lva1-app6892/85/Modellistica-molecolare-e-applicazioni-alla-sclerosi-multipla-15-320.jpg)
![PDB 3RDT Crystal Structure of 809.B5 TCR + MHC Class II I-Ab/3k peptide
TCR : [peptide : MHC] Struttura Cristallografica
T Cell
peptide
MHC
Tasca di
legame](https://image.slidesharecdn.com/pieroni-150724112126-lva1-app6892/85/Modellistica-molecolare-e-applicazioni-alla-sclerosi-multipla-16-320.jpg)