Stella Colomba
- 1. Universit├Ā di Urbino ŌĆ£Carlo BoŌĆØ Dip.to Scienze della Terra, della Vita e dellŌĆÖAmbiente (DiSTeVA) Dott.ssa M. Stella Colomba Dott. Armando Gregorini Analisi dei profili di espressione dei geni coinvolti nel metabolismo dellŌĆÖacido arachidonico in pazienti affetti da depressione maggiore o disturbo bipolare Expression profiling of genes involved in arachidonic acid metabolism in patients with major depression or bipolar disorder
- 2. Obiettivo dello studio (Aims) Analisi dei profili di espressione dei geni coinvolti nella sintesi e nel metabolismo dellŌĆÖacido arachidonico in soggetti con depressione maggiore o disturbo bipolare per investigare: ’ü¼ le cause dellŌĆÖaccumulo di acido arachidonico nella membrana piastrinica; ’ü¼ potenziali geni da utilizzare come biomarker nella diagnosi dei disturbi dellŌĆÖumore.
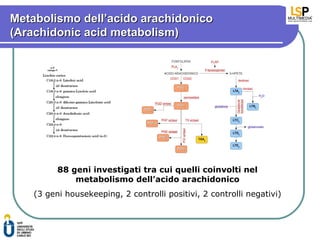
- 3. Metabolismo dellŌĆÖacido arachidonico (Arachidonic acid metabolism) 88 geni investigati tra cui quelli coinvolti nel metabolismo dellŌĆÖacido arachidonico (3 geni housekeeping, 2 controlli positivi, 2 controlli negativi)
- 4. Campione (Subjects) ’ü¼ Pazienti con depressione maggiore n=5 ’ü¼ Pazienti con disturbo bipolare n=5 ’ü¼ Controlli n=5 ’ü¼ Prelievo ematico in EDTA e isolamento dei linfociti circolanti.
- 5. Metodo: Saggi in Real-Time PCR (Method: Real-Time PCR assays) ’ü¼ Tecnica standardizzata, di facile realizzazione ed adatta agli studi di routine di biologia molecolare. ’ü¼ Metodo altamente sensibile ed affidabile per la valutazione e/o quantificazione simultanea nello stesso campione dellŌĆÖespressione genica di numerosi geni (sia rari che abbondanti).
- 6. Procedura sperimentale (Real-Time PCR work flow) Purify RNA from Prepare cDNA from Add cDNA to Aliquot mix into the plate lymphocytes purified RNA Mastermix Cycle in real-time cycler Analyse results
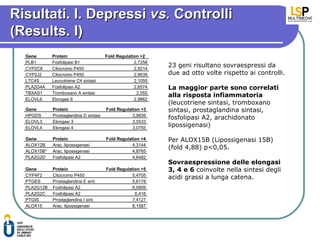
- 7. Risultati. I. Depressi vs. Controlli (Results. I) Gene Protein Fold Regulation >2 PLB1 Fosfolipasi B1 2,7258 CYP2C8 Citocromo P450 2,9214 23 geni risultano sovraespressi da CYP2J2 Citocromo P450 2,9639 due ad otto volte rispetto ai controlli. LTC4S Leucotriene C4 sintasi 2,1055 PLA2G4A Fosfolipasi A2 2,6574 La maggior parte sono correlati TBXAS1 Tromboxano A sintasi 2,055 alla risposta infiammatoria ELOVL6 Elongasi 6 2,9862 (leucotriene sintasi, tromboxano Gene Protein Fold Regulation >3 sintasi, prostaglandina sintasi, HPGDS Prostaglandina D sintasi 3,9839 fosfolipasi A2, arachidonato ELOVL3 Elongasi 3 3,5533 ELOVL4 Elongasi 4 3,0755 lipossigenasi) Gene Protein Fold Regulation >4 Per ALOX15B (Lipossigenasi 15B) ALOX12B Arac. lipossigenasi 4,3144 (fold 4,88) p<0,05. ALOX15B* Arac. lipossigenasi 4,8765 PLA2G2D Fosfolipasi A2 4,6482 Sovraespressione delle elongasi Gene Protein Fold Regulation >5 3, 4 e 6 coinvolte nella sintesi degli CYP4F2 Citocromo P450 5,4705 acidi grassi a lunga catena. PTGES Prostaglandina E sint. 5,6178 PLA2G12B Fosfolipasi A2 6,0909 PLA2G2C Fosfolipasi A2 6,416 PTGIS Prostaglandina I sint. 7,4127 ALOX15 Arac. lipossigenasi 8,1587
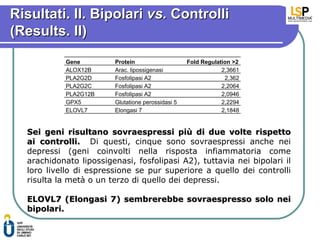
- 8. Risultati. II. Bipolari vs. Controlli (Results. II) Gene Protein Fold Regulation >2 ALOX12B Arac. lipossigenasi 2,3661 PLA2G2D Fosfolipasi A2 2,362 PLA2G2C Fosfolipasi A2 2,2064 PLA2G12B Fosfolipasi A2 2,0946 GPX5 Glutatione perossidasi 5 2,2294 ELOVL7 Elongasi 7 2,1848 Sei geni risultano sovraespressi pi├╣ di due volte rispetto ai controlli. Di questi, cinque sono sovraespressi anche nei depressi (geni coinvolti nella risposta infiammatoria come arachidonato lipossigenasi, fosfolipasi A2), tuttavia nei bipolari il loro livello di espressione se pur superiore a quello dei controlli risulta la met├Ā o un terzo di quello dei depressi. ELOVL7 (Elongasi 7) sembrerebbe sovraespresso solo nei bipolari.
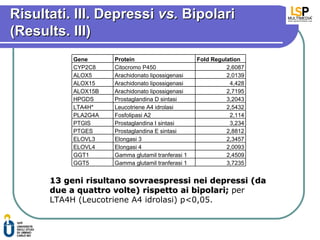
- 9. Risultati. III. Depressi vs. Bipolari (Results. III) Gene Protein Fold Regulation CYP2C8 Citocromo P450 2,6087 ALOX5 Arachidonato lipossigenasi 2,0139 ALOX15 Arachidonato lipossigenasi 4,428 ALOX15B Arachidonato lipossigenasi 2,7195 HPGDS Prostaglandina D sintasi 3,2043 LTA4H* Leucotriene A4 idrolasi 2,5432 PLA2G4A Fosfolipasi A2 2,114 PTGIS Prostaglandina I sintasi 3,234 PTGES Prostaglandina E sintasi 2,8812 ELOVL3 Elongasi 3 2,3457 ELOVL4 Elongasi 4 2,0093 GGT1 Gamma glutamil tranferasi 1 2,4509 GGT5 Gamma glutamil tranferasi 1 3,7235 13 geni risultano sovraespressi nei depressi (da due a quattro volte) rispetto ai bipolari; per LTA4H (Leucotriene A4 idrolasi) p<0,05.
- 10. Conclusioni (Conclusions) ’ü¼ Premesso che i geni candidati potenzialmente associati alla depressione maggiore o al disturbo bipolare dovranno essere ulteriormente validati attraverso lŌĆÖanalisi di un elevato numero di pazienti, i dati ottenuti, sebbene ancora preliminari, sembrano indicare che la maggior parte dei geni sovraespressi ├© correlata alla risposta infiammatoria che caratterizza i disturbi dellŌĆÖumore e, in particolare, la depressione maggiore. ’ü¼ La sovraespressione differenziata delle elongasi (enzimi responsabili della sintesi di acidi grassi a lunga catena) nei soggetti affetti da depressione maggiore o disturbo bipolare risulta essere di particolare interesse e merita ulteriori approfondimenti.