DNA_fingerprinting.......................
- 1. Sun Elena 4E 1 di 8 RELAZIONE DI LABORATORIO TITOLO: DNA Fingerprinting (Test di paternità ) OBIETTIVO: Confrontare due o più DNA per confermare o escludere l'esistenza di un rapporto di parentela PREMESSA: Il DNA fingerprinting (o impronta genetica o DNA profiling) è una tecnica ampiamente usata, dall'ambito medico a quello forense, per l'identificazione di un soggetto. Il DNA fingerprinting si basa sull'analisi di marcatori STR (Short Tandem Repeats o microsatelliti) presenti nel genoma di ogni individuo. Gli STR consistono in brevi sequenze di DNA (10-50 paia di basi) ripetute in tandem numerose volte e presenti in diversi cromosomi. Il numero di ripetizioni è altamente variabile nei diversi individui, ma è costante in ogni profilo e segue un pattern di ereditarietà mendeliana. L'analisi di sequenze STR può essere effettuata mediante il taglio del DNA con enzimi di restrizione (particolari proteine capaci di tagliare la molecola del DNA in siti specifici). Si ottengono in questo modo frammenti di DNA di diversa lunghezza, contenenti sequenze STR, che possono essere separati tramite elettroforesi su gel di agarosio. L’elettroforesi è una tecnica che permette di separare frammenti di acido nucleico, in funzione del loro peso molecolare. Frammenti di DNA (carichi negativamente per la presenza dei gruppi fosfato) in un campo elettrico migrano verso il polo positivo e migrano tanto più velocemente quanto più sono piccoli. In questo modo dal DNA è possibile ottenere un profilo genetico specifico per ciascun individuo e quindi confrontare il profilo genetico tra i possibili padri dell’individuo. CENNI TEORICI: • Cosa sono gli STR? I marcatori microsatelliti STR (Short Tandem Repeat) sono delle sequenze di DNA composte da quattro nucleotidi ripetuti in tandem, che hanno un elevato grado di polimorfismo nella popolazione umana. In parole più semplici, nella nostra sequenza di DNA ci sono delle regioni con una “lunghezza variabile” da individuo a individuo. Andando a identificare molte di queste regioni possiamo determinare un profilo genetico unico per ogni essere umano. Essendo discriminanti, misurabili e invariabili nel tempo vengono scelti come marcatori per distinguere gli individui gli uni dagli altri. • Cosa sono gli enzimi di restrizione? Gli enzimi di restrizione sono particolari enzimi in grado di riconoscere delle sequenze specifiche di nucleotidi lungo un filamento di DNA e di catalizzare un taglio (un'idrolisi) esattamente in
- 2. Sun Elena 4E 2 di 8 corrispondenza di esse. La loro scoperta è avvenuta mediante l'osservazione che il DNA proveniente da un ceppo di Escherichia coli se introdotto (tramite trasformazione batterica) in un ceppo diverso, veniva letteralmente tagliato in piccoli frammenti. In seguito, si osservò che la stessa cosa accadeva al DNA virale quando i batteri venivano infettati. Ovvero, il DNA del virus veniva fatto a pezzettini e questi frammenti erano così resi inattivi. Si disse che la crescita virale era ristretta in un determinato ceppo batterico, da qui il termine "restrizione" per definire gli enzimi in grado di fare quanto osservato. In particolare, i batteri possiedono quelle che vengono definite endonucleasi di restrizione, enzimi che tagliano i legami interni ad una certa sequenza nucleotidica sia essa di un filamento di DNA o all'interno di DNA circolare. • Che cos’è l’elettroforesi? L'elettroforesi è una tecnica utilizzata nell'ambito delle analisi di laboratorio che sfrutta la massa molecolare e la carica elettrica delle proteine, per valutarne la quantità e la qualità . In particolare, quest'esame consente di separare le proteine in cinque frazioni: albumina, alfa 1 globuline, alfa 2 globuline, beta globuline e gamma globuline. L'alterazione del rapporto tra questi tipi di proteine è indicativa di alcune condizioni patologiche. L'elettroforesi può essere eseguita su campioni di siero sanguigno, urine o altri liquidi biologici, come il liquor (liquido cerebrospinale).
- 3. Sun Elena 4E 3 di 8 MATERIALI E STRUMENTI: 1. Strumentazione occorrente Camera per elettroforesi orizzontale con vassoio, pettine e gel agarosio Micropipette da 10-100 µl (per prelevare i campioni di DNA) Alimentatore per elettroforesi Puntali adatti alle micropipette 2. Materiali 4 provette (eppendorf) con DNA e loading DYE (LD): 1. Della madre 2. Del figlio 3. Del possibile padre #1 4. Del possibile padre #2 Fast Blast DNA stain a concentrazione 100x (è il colorante che permette di visualizzare la corsa elettroforetica senza aver bisogno del trans illuminatore); Pipetta graduata da 10mL Becker 250mL
- 4. Sun Elena 4E 4 di 8 TAE 1X (Tris-Acetato EDTA) (è un buffer con potere tamponante e che conduce corrente elettrica) Acqua distillata Cilindro graduato PROCEDURA: 1. Preparazione del gel d'agarosio • Utilizzando la vaschetta per elettroforesi inserire gli appositi pettini per la formazione dei pozzetti. • Sciogliere il gel di agarosio contenuto nel flacone in un forno a microonde o su una piastra riscaldante, togliere il tappo e mescolare dopo un minuto a 550W. • Quando la soluzione sarà completamente trasparente, attendere qualche minuto per farla raffreddare fino a circa 50°. • Versare il gel nell’apposita vaschetta con pettini, facendo attenzione a non formare bolle • Aspettare circa 30 minuti perché raffreddandosi il gel polimerizza. • Una volta che il gel è polimerizzato, togliere il pettine per la formazione dei pozzetti e ricoprire il gel con il tampone TAE buffer 1x. Per ottenerlo prelevare 50ml di buffer concentrato 5X in 200ml di acqua distillata. 2. Preparazione dei campioni e corsa elettroforetica
- 5. Sun Elena 4E 5 di 8 • Aggiungere 10 μl di ciascun campione nei pozzetti utilizzando una micropipetta. Pozzetto1- DNA della madre Pozzetto2- DNA del bambino Pozzetto3- DNA del possibile padre #1 Pozzetto4- DNA del possibile padre #2 • Chiudere il coperchio dell’alimentatore, accendere l’alimentatore e verificare il passaggio di corrente attraverso l’emissione di bolle in prossimità degli elettrodi. La corsa elettroforetica necessita di un voltaggio di circa 12-15 V/cm. Per un gel di 8 cm di lunghezza il voltaggio è di 100 V. • Quando il fronte di migrazione indicato dal Loading Dye (LD) di colore blu raggiunge la fine del gel (circa 90 minuti) staccare l’alimentatore e procedere alla colorazione per la visualizzazione delle bande. 3. Colorazione del gel mediante Fast Blast DNA Stain • Una volta arrestata la corsa elettroforetica, estrarre il gel e immergerlo in una vaschetta contenete il colorante del DNA ad una concentrazione di 1x per circa 20-30 minuti, agitando lentamente • Trasferire il gel in un nuovo contenitore per la decolorazione • Decolorare il gel mediante due lavaggi in acqua calda di 5 minuti ciascuno, sempre agitando lentamente e con cautela OSSERVAZIONE e CONCLUSIONE:
- 6. Sun Elena 4E 6 di 8 Osservare dopo un po’ la comparsa delle bande. Confrontare il profilo genetico del figlio con i due possibili padri. Il profilo genetico del figlio è combaciante con il profilo genetico del secondo padre. Che cos’è la PCR? La reazione a catena della polimerasi, in inglese Polymerase Chain Reaction o PCR, è una tecnica di biologia molecolare inventata nel 1983 che permette di replicare in maniera selettiva degli specifici tratti di DNA. Questo processo, che avviene in natura all'interno di tutte le cellule viventi, viene riprodotto in vitro in ambiente controllato e applicato in campo medico e diagnostico, forense e nelle analisi alimentari. La PCR funziona un po' come una fotocopiatrice che, partendo da un segmento di DNA e conoscendone il punto di inizio e di fine, ne riesce a produrre più copie identiche in breve tempo. Il primo step per effettuare la PCR è quello di misurare e mixare con un ordine preciso tutti i reagenti necessari all'interno di una provetta. Questa sarà poi inserita in un macchinario chiamato termociclatore. Nello specifico gli ingredienti che ci serviranno sono: • DNA originale/bersaglio: deve essere preventivamente estratto e purificato per essere utilizzato come stampo; • DNA polimerasi termostabile: si tratta di un enzima fondamentale, senza il quale non potrebbe avvenire la replicazione; • Primers: sono brevi frammenti di DNA sintetici che faranno partire e terminare la reazione di amplificazione; • Desossiribonucleotidi trifosfato (dNTP): hanno un nome complicato ma sono semplicemente i "mattoncini" – a cui però è stato aggiunto uno zucchero e un gruppo fosfato- che verranno aggiunti, uno per uno e sempre in una direzione specifica, per riformare la catena complementare di DNA; • Soluzione buffer: si tratta di una soluzione contenente additivi che mantiene il pH di reazione stabile; • Altri additivi: migliorano le prestazioni della reazione; • Acqua distillata: la si aggiunge per portare il mix al volume desiderato.
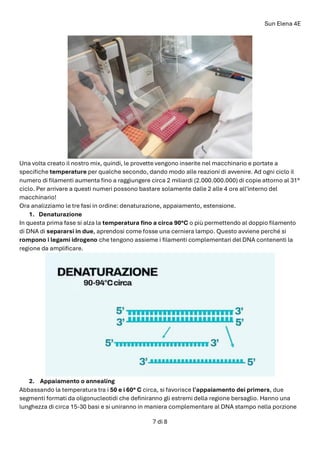
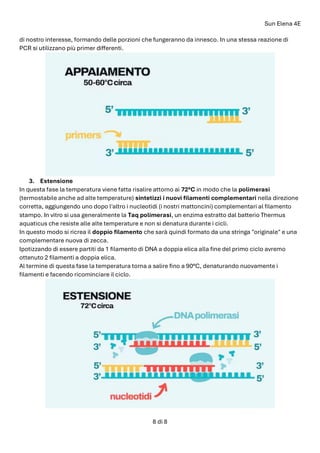
- 7. Sun Elena 4E 7 di 8 Una volta creato il nostro mix, quindi, le provette vengono inserite nel macchinario e portate a specifiche temperature per qualche secondo, dando modo alle reazioni di avvenire. Ad ogni ciclo il numero di filamenti aumenta fino a raggiungere circa 2 miliardi (2.000.000.000) di copie attorno al 31° ciclo. Per arrivare a questi numeri possono bastare solamente dalle 2 alle 4 ore all'interno del macchinario! Ora analizziamo le tre fasi in ordine: denaturazione, appaiamento, estensione. 1. Denaturazione In questa prima fase si alza la temperatura fino a circa 90°C o più permettendo al doppio filamento di DNA di separarsi in due, aprendosi come fosse una cerniera lampo. Questo avviene perché si rompono i legami idrogeno che tengono assieme i filamenti complementari del DNA contenenti la regione da amplificare. 2. Appaiamento o annealing Abbassando la temperatura tra i 50 e i 60° C circa, si favorisce l'appaiamento dei primers, due segmenti formati da oligonucleotidi che definiranno gli estremi della regione bersaglio. Hanno una lunghezza di circa 15-30 basi e si uniranno in maniera complementare al DNA stampo nella porzione
- 8. Sun Elena 4E 8 di 8 di nostro interesse, formando delle porzioni che fungeranno da innesco. In una stessa reazione di PCR si utilizzano più primer differenti. 3. Estensione In questa fase la temperatura viene fatta risalire attorno ai 72°C in modo che la polimerasi (termostabile anche ad alte temperature) sintetizzi i nuovi filamenti complementari nella direzione corretta, aggiungendo uno dopo l'altro i nucleotidi (i nostri mattoncini) complementari al filamento stampo. In vitro si usa generalmente la Taq polimerasi, un enzima estratto dal batterio Thermus aquaticus che resiste alle alte temperature e non si denatura durante i cicli. In questo modo si ricrea il doppio filamento che sarà quindi formato da una stringa "originale" e una complementare nuova di zecca. Ipotizzando di essere partiti da 1 filamento di DNA a doppia elica alla fine del primo ciclo avremo ottenuto 2 filamenti a doppia elica. Al termine di questa fase la temperatura torna a salire fino a 90°C, denaturando nuovamente i filamenti e facendo ricominciare il ciclo.