Genpop9coal e abc
- 1. Programma del corso 1. Diversità genetica 2. Equilibrio di Hardy-Weinberg 3. Unione assortativa 4. Linkage disequilibrium 5. Mutazione 6. Deriva genetica 7. Flusso genico e varianze genetiche 8. Selezione 9. Mantenimento dei polimorfismi e teoria neutrale 10. Introduzione al coalescente 11. Evidenze genetiche sull’evoluzione umana
- 2. Test di selezione: Tajima In assenza di ricombinazione, se un locus si è evoluto in condizioni di neutralità, due statistiche (numero di siti polimorfici; mismatch medio) sono entrambi funzione di 4Nμ. Il test di Tajima calcola la differenza, D, fra due stime di 4Nμ (basate rispettivamente su numero di siti polimorfici e mismatch medio) e la confronta con una distribuzione casuale. In una popolazione all’equilibrio fra deriva e mutazione E(π) = a1 4Nμ E(k) = 4Nμ Se S è il numero di siti polimorfici, e K il mismatch medio S/ a1 = K, e d = K – (S/ a1) La statistica di Tajima è D = d / Var(d)
- 4. Test di selezione: Tajima D<0 Eccesso di siti polimorfici: selezione diversificante D>0 Eccesso di mismatch: selezione stabilizzante
- 5. Un esempio. DNA di 5 individui Position 12345 67890 12345 67890 Person Y 00000 00000 00000 00000 Person A 00100 00000 00100 00010 Person B 00000 00000 00100 00010 Person C 00000 01000 00000 00010 Person D 00000 01000 00100 00010
- 6. You vs A Person Y 00000 00000 00000 00000 Person A 00100 00000 00100 00010 3 polymorphisms You vs B Person Y 00000 00000 00000 00000 Person B 00000 00000 00100 00010 2 polymorphisms You vs C Person Y 00000 00000 00000 00000 Person C 00000 01000 00000 00010 2 polymorphisms And so on, for 10 comparisons
- 7. The average number of polymorphisms is And the number of polymorphic sites is 4: Position 12345 67890 12345 67890 Person Y 00000 00000 00000 00000 Person A 00100 00000 00100 00010 Person B 00000 00000 00100 00010 Person C 00000 01000 00000 00010 Person D 00000 01000 00100 00010 d=2‚àí4=‚àí2
- 8. Un problema col test di Tajima past present Expanding population Stationary population
- 9. Nucleotide substitutions in the chromosomes of different individuals Only a few are shared Many are shared
- 10. Distributions of pairwise sequence differences, or mismatch distributions Unimodal Multimodal Peak ◄► age of expansion Many peaks Tajima’s D < 0 Tajima D >0 (= diverisfying selection) (= stabilizing selection)
- 11. La genetica studia la trasmissione ereditaria dal passato al presente forward
- 12. Ma quando si lavora su popolazioni si raccolgono dati sul presente e si cerca di risalire al passato ? ? backward
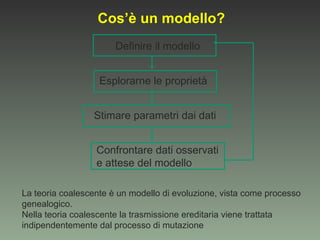
- 13. Cos’è un modello? Definire il modello Esplorarne le proprietà Stimare parametri dai dati Confrontare dati osservati e attese del modello La teoria coalescente è un modello di evoluzione, vista come processo genealogico. Nella teoria coalescente la trasmissione ereditaria viene trattata indipendentemente dal processo di mutazione
- 14. Costruiamo (procedendo verso il passato) la genealogia materna di un gruppo di individui Due possibilità: o ogni individuo ha una madre diversa: O due individui hanno la stessa madre: Chiamo questo fenomeno coalescenza
- 15. Assunzioni del coalescente classico (Kingman 1982) 1. Neutralità 2. Siti infiniti 3. Se gli individui sono diploidi e le dimensioni della popolazione sono N, il modello vale per 2N copie aploidi e indipendenti del gene 4. Unione casuale entro la popolazione 5. Dimensioni della popolazione costanti (*) 6. Generazioni non sovrapposte Parliamo di caratteri a trasmissione uniparentale
- 16. Ricostruire la storia di una popolazione Passato Presente
- 17. Genealogie N = 10 N costante n=6 9 generazioni
- 18. Genealogie MRCA
- 19. Genealogie MRCA
- 20. Mutazione
- 21. CAATG Mutazione CAATA CAGTG TAATA CGGTG 1 1 2 3 3 TAACA 4 5 CAATG CGGTG CAGTG CAGTG TAACA TAATA
- 22. Non sempre l’albero ricostruito sulla base delle mutazioni è molto informativo 2 3 1 4 5 CGGTG CAGTG CAATG TAATA TAACA Possiamo capire qualcosa di più?
- 23. Nel risalire dal presente al passato incontriamo una successione di eventi di coalescenza. Conseguenze: 1. In un campione di r individui alla generazione 0, il numero di antenati 1, 2,…n generazioni fa (ξ1, ξ2, ..ξn) decresce fino ad arrivare a 1: r = ξ0 ≥ ξ1 ≥ ξ2 ≥, ..ξn Ogni genealogia viene ricondotta necessariamente a un singolo antenato comune (MRCA). Non è possibile discriminare fra monofilia e polifilia
- 24. Nel risalire dal presente al passato incontriamo una successione di eventi di coalescenza. Conseguenze: N1=8 ? N0=8 2. Se la popolazione è stazionaria (N costante), N donne hanno una madre fra le N donne della generazione precedente. La probabilità di coalescenza è vicina a 1/N
- 25. Nel risalire dal presente al passato incontriamo una successione di eventi di coalescenza. Conseguenze: campione P(ant.com) P(n) 2 1/N 1-(1/N) 3 ≈2/N 1-(2/N) n n /N 1- n /N 2 2 3. La probabilità P(n) che n alleli abbiano n antenati distinti alla generazione precedente diminuisce con le dimensioni del campione (più grande il campione, più grande la P di almeno un evento di coalescenza)
- 26. Dimensioni effettive AA AA 4. La dimensione YX XX effettiva della mt mt popolazione è proporzionale a: NC = 2 AA AA NeA = 4 NeX = 3 YX XX NeY = 1 mt mt Ne mt = 1
- 27. Nel risalire dal presente al passato incontriamo una successione di eventi di coalescenza. Conseguenze: 5. I tempi medi di coalescenza aumentano procedendo verso il passato
- 28. Nel risalire dal presente al passato incontriamo una successione di eventi di coalescenza. Conseguenze: Tempo atteso per passare da k a (k-1) antenati: Tk = 4N/[k(k-1)], o 2N per geni a trasm. uniparentale 6. Il tempo atteso fra due eventi di coalescenza è distribuito esponenzialmente. E(T)=4N: Wright-Fisher
- 29. Come si può arricchire il coalescente classico? 1. Selection 2. Recurrent and back mutation 3. Recombination 4. *Non-random mating: eg geographic subdivision with specified migration between subpopulations 5. Population size fluctuation, including bottlenecks and expansions 6. Non-’Poisson’ distributions of offspring numbers 7. Unequal generation intervals between lineages
- 30. Utilizzo del coalescente: simulazione di processi genetici complicati • Per capire la demografia di popolazioni passate (colli di bottiglia, espansioni...) • Per stimare il momento più probabile in cui sono avvenute mutazioni, migrazioni, cambiamenti delle pressioni selettive... • Per valutare se i dati disponibili sono sufficienti a discriminare fra diverse ipotesi
- 31. ABC (Approximate Bayesian Calculations) 1. Alternative models are defined 2. For each model, millions of genealogies are generated by coalescent simulations, sampling parameters from broad prior distributions 3. Summary statistics are estimated from observed and simulated data 4. A subset of simulations is retained, i.e. those showing the closest correspondence between observed and simulated statistics 5. MODEL CHOICE: The posterior probability of each model is evaluated by counting its occurrences among the best simulations 6. PARAMETER ESTIMATION: The modes are calculated of the parameter values producing the best simulations under the chosen model, and credible intervals about the modes are estimated
- 32. ABC (Approximate Bayesian Computations) 1. Alternative models are defined Mod 1 Mod 2 Mod 3 E a1 a2 a1 a2 E E M M M 27 27 27 26 26 26 C C C
- 33. ABC (Approximate Bayesian Computations) 2. For each model, millions of genealogies are generated, sampling parameters from broad prior distributions Parameters Priors Ne Modern Tuscans 50 000 – 500 000 μ 0.0003 – 0.0075 T estimated (bottleneck) 101 – 1500 Ne Generation 26 100 – 10 000 Ne Generation 27 10 000 – 100 000 Ne at split 100 – 2000 Ne Medieval Tuscans 10 000 – 50 000 Ne Etruscans 4000 – 21 000
- 34. ABC (Approximate Bayesian Computations) 3. A subset of simulations is retained, i.e. those with the shortest distances between observed and simulated statistics Observed statistics
- 35. ABC (Approximate Bayesian Computations) 4. MODEL CHOICE: The posterior probability of each model is estimated from the frequency among the best simulations of the simulations generated under that model Observed statistics P=0.70 Mod 1 P=0.10 P=0.20 E a1 a2 a1 a2 E E M M M 27 27 27 26 26 26 C C C
- 36. ABC (Approximate Bayesian Computations) 5. PARAMETER ESTIMATION: the mode and the highest probability density interval of the parameters are estimated from the best simulations under the best model Observed statistics μ 100 Ne generation 0 T bottleneck Ne Generation 26 40 Ne Generation 27 27 26 Ne generation 40 0 Ne generation 100
- 38. Filogeografia: Y
- 40. Filogeografia: di male in peggio
- 41. Gene trees, population trees Gene trees are unknown, but we can reconstruct some of their features assuming mutations occurred at a constant rate (=no selection)
- 42. Population-genetics theory describes the expected features of gene trees in terms of population parameters T2=2N 4N T5=N/5 For nuclear genes, Exp time from k to (k-1) ancestors: Tk = 4N/[k(k-1)] generations (large std. errors)
- 43. If two populations are isolated, the final coalescence is 2N generations before the split past Exp (T) = 4N Exp(T2) = 2N Exp τ = T/2 T: gene divergence T2=2N Origin of the B population A B τ: population divergence present
- 44. If there is initial polymorphism, the final coalescence may be much more than 2N generations before the split past T>> τ T2=2N Origin of the B population A B present
- 45. Gene divergence predates population divergence: T is equal to τ only if 2 Ne =0 past E(T-τ) = 2 Ne T: gene generations divergence τ: population divergence present
- 46. Only if there is a population bottleneck or a founder effect does τ approximate T past Phylogeographic analyses require the 2Ne very small assumption of strong founder effects τ≈T present
- 47. Is it safe to assume that most human populations originated from a founder effect? Evidence for rapid expansion (110-40 Kyrs ago) in farming populations Evidence for shrinking in hunting-gathering populations, possible caused by competition with early farmers (Excoffier & Schneider 1999) Necessary to test for founder effects causing reduced genetic diversity at several loci in a population
- 48. Ages of molecules are not ages of populations Initial polymorphism results in overestimation of the population’s age Population’s age Coalescence time From Krings et al. (1997)
- 49. Any conclusions? • Robust inferences on past population processes are complicated • No shortcuts: allele genealogies are not population genealogies • Archaeologists need geneticists to tell demographic from cultural processes • Geneticists need archeologists to identify good hypotheses to test
- 50. Sintesi 1 • Il coalescente fornisce un modello di evoluzione basato sull’indipendenza fra processo genealogico e processo mutazionale • Tramite il coalescente si possono stimare parametri sulla base di assunzioni esplicite e simulare geenalogie di geni
- 51. Sintesi 2 Vantaggi del coalescente: • Rende esplicite le assunzioni; • fornisce misure di incertezza; • non tratta (a differenza degli approcci filogeografici) i polimorfismi come mutazioni fissate.



























![Nel risalire dal presente al passato incontriamo
una successione di eventi di coalescenza.
Conseguenze:
Tempo atteso per passare da k a (k-1) antenati:
Tk = 4N/[k(k-1)], o 2N per geni a trasm. uniparentale
6. Il tempo atteso fra due eventi di coalescenza è
distribuito esponenzialmente. E(T)=4N: Wright-Fisher](https://image.slidesharecdn.com/genpop9coaleabc-120604025911-phpapp02/85/Genpop9coal-e-abc-28-320.jpg)













![Population-genetics theory describes
the expected features of gene trees in
terms of population parameters
T2=2N
4N
T5=N/5
For nuclear genes, Exp time from k to (k-1) ancestors:
Tk = 4N/[k(k-1)] generations (large std. errors)](https://image.slidesharecdn.com/genpop9coaleabc-120604025911-phpapp02/85/Genpop9coal-e-abc-42-320.jpg)
































































